Hoy día 27 de Junio de 2011 ha empezado el proyecto Praktikum.
 (Politécnica de la Universitat de Valencia)
(Politécnica de la Universitat de Valencia)
En mi caso, me confundí de lugar en el que se nos había citado, pero no pasó nada ya que pronto fuimos (yo y mi tutora, la profesora de biología) al lugar indicado, llegando con cinco minutos de adelanto. Estaba bastante nervioso ya que era una gran experiencia nueva para mí.
En dicho punto de encuentro se nos informó, breve y generalemente, de los grados que se podían cursar en la politécnica, más concretamente aquellas que se podían cursar en la escuela de agronomía y del medio natural, que son la ingeniería forestal, la rural, la tecnología y manipulación de alimentos y la biotecnología. Tras la charla, cada alumno fue con su respectivo tutor para iniciar el proyecto que se le había asignado.
En mi caso, yo soy Diego, y mi trabajo consistía, junto con Borja y bajo la supervisación del tutor Gonzalo, en analizar y estudiar los microorganismos productores de antibióticos.
Al inicio del proyecto, se nos envió al laboratorio de Microbiología, del departamento de Biotecnología. Una vez allí, se nos presentaron las diferentes herramientas de trabajo que íbamos a necesitar: los matraces, las placas, los medios y productos de cultivo, el autoclave,… Que explicaré a lo largo del blog conforme utilize dichas herramientas.
Lo primero era preparar los medios de cultivo: para ello necesitábamos matraces simples, agár (mezclado con nutrientes para los microorganismos), agua, algodón y aluminio. Se trataba de verter agár en el matráz y mezclarlo con agua, en una proporción de 38g por cada 1L de agua; puesto que utilizamos 300ml de agua, utilizamos 11.4g de agár (el agár es una sustancia sólida que se funde a 100ºC, pero que se solidifica a 45ºC. Por ello lo utilizámos para obtener un medio de cultivo sólido posteriormente). Después cubríamos el matráz con el algodón (que no es como el que tú tendrás en casa) y lo volvíamos a cubrir con aluminio. Esta operación sirviría para, tiempo después, tener medios de cultivo en los que colocar los microorganismos.
El matraz se colocaba en una máquina llamada autoclave, que según Gonzalo, es como «una gran olla a presión»: se colocaban los matraces dentro, la máquina los calentaba hasta los 21º durante 20 minutos, y así se esterilizaban los matraces. El autoclave expulsaba 75 L de vapor de agua para empujar y expulsar a su vez los gases que pudiera contener el recipiente. Después, salía agua muy caliente que se encargaba de eliminar los microorganosmos que pudiran estar presentes en los matraces. Todo esto tardaba, junto al proceso de enfriamiento, 1h y 15 min, tiempo que aprovechamos para almorzar en una de las numerosas cafeterías de la politécnica.
 (autoclave)
(autoclave)  (así es como se colocan en el autoclave)
(así es como se colocan en el autoclave)
Al volver, Gonzalo nos dió unos medios de cultivo que ya estaban preparados: por un lado, teníamos unas placas petri sin microorganismos (solo con em medio sólido y los nutrientes), y por otro lado, teníamos placas petri con microorganismos que ya habían proliferado. Teníamos que obtener microorganismos de esas placas de manera que estubiesen bien separados. Para ello, cogimos un asa, un mechero (de los gordos, los de laboratorio) y las placas. Primero, esterilizamos el asa colocándolo un tiempo sobre el mechero (encendido, obviamente), después, con dicho asa, cogíamos microorganismos de una de las placas y los repartíamos en un lado de una placa petri sin microorganismos; esto era la primera estría. Luego, volvíamos a esterilizar el asa y repartíamos los microorganismos de la primera estría en una segunda estría, dejando un hueco para poder posteriormente hacer una tercera estría de microorganismos a partir de la segunda, habiendo esterilizado el asa anteriormente. Dejamos esas nuevas cajas petri con microorganismos en un gran recipiente que se mantenía a 28 ºC, recogimos nuestros cultivos que habíamos dejado en el autoclave, para después vertir dichos cultivos (que estaban a 50 ºC) en unas cajas petri vacías y las guardamos en otro recipiente. Para hoy, el primer día, ya era suficiente.
 (medio de cultivo)
(medio de cultivo)  (asa de siembra)
(asa de siembra)
Mientras estábamos en el laboratorio, me gustó mucho ver que estábamos en medio de un grupo de verdaderos profesionales y cómo nos ayudaban en el proyecto. Me impresionó mucho la gran cautela con la que se manejaban los instrumentos, asi como la gran cantidad de reglas higiénicas que había que cumplir en el laboratorio.
Tras el proyecto, nos fuimos a comer en grupo a un establecimiento situado en la parte posterior del edificio en el que trabajamos, y después nos fuimos a una aula de informática donde estoy escribiendo ahora mismo.
Al acabar el horario previsto, me voy al colegio mayor Galileo Galilei, ya que al ser de Alicante no me puedo permitir el lujo de volver a mi casa 🙁
El segundo día, hemos empezado las prácticas directamente, a las 9:15 de la mañana, en el laboratorio. Nos han enseñado cómo hacer una tinción Gram, para colorar micoorganismos (aunque mueran) y poder examinarlos luego por microscopio. Si en el resultado salía un color violeta oscuro, entonces el microorganismo es Gram- positivo (su pared bacteriana es gruesa, y por tanto retiene mejor los colorantes), y si da un color rosa claro, entonces el microorganismo es Gram-negativo (su pared bacteriana es fina). El procedimiento fue el siguiente:
Colocamos sobre una gota de agua destilada microorganismos provenientes de una placa petri en una placa de vidrio mediante un asa convenientemente esterilizada. Después, añadíamos violeta de genciana a la placa, sobre los microorganismos, y esperamos un minuto. Después, lavamos la placa y le colocamos encima lugól, para que el anterior colorante se fije bien a la pared bacteriana. Tras un minuto, se vuelve a lavar (siempre con agua destilada) y le echamos alcohol durante 30 segundos (hay que tener mucho cuidado con este paso, ya que si nos pasamos de tiempo se pueden deformar los resultados). Luego lavamos la placa y le echamos fuscina básica, que se tiene que mantener durante tres minutos. Borja y yo, que somos unos bestias, hemos hecho cuatro placas a la vez, y yo me he manchado el dedo pulgar con violeta de genciana, que resulta que no se fué con lejía, por lo que tendré que esperar varios días para que se me vaya.
Tras esto, secamos las placas usando un mechero (de los de laboratoooooorio). Y así, las placas estaban listas para ser observadas por microscopio. Primero, observamos a 40 aumentos, pero resultó que no era suficiente, teníamos que usar 100 aumentos. A esos aumentos teníamos que usar un aceite de inmersión que englobase en una gota el objetivo y la placa, ya que a esos aumento se debe evitar el índice de refracción. Al ver a los microorganismos, observamos que se dividían en dos tipos: unos microorganismos tenían forma redondeada (que son los llamados cocos) y otros tenían forma de tubo (que son los bacilos).
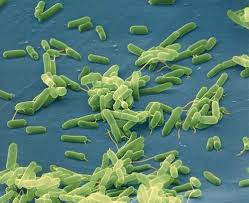 (bacilos)
(bacilos) 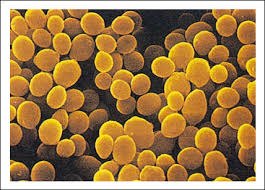 (cocos)
(cocos)
Después de observar las placas, nos tomamos un buen almuerzo en la cafetería del día anterior. Al volver, Gonzalo nos dió una charla muy interesante sobre los proyectos que se estaban realizando en el departamento de Biotecnología.
 (almuerzo)
(almuerzo)
Resulta que tiene varias líneas de investigación, siendo una de ellas el estudio de microorganismos que pudieran descomponer las aguas residuales. Este proyecto tiene un fuerte apoyo financiero otorgado por los ayuntamientos comaracales (y también del general), ya que les interesa mucho encontrar organismos que puedan descomponer totalmente los resíduos de estas aguas. Otra investigación que están haciendo está relacionada con los fungicidas. Se buscan, a nivel internacional, fungicidas naturales, ya que los sintéticos (los preductos químicos) contaminan muchísimo, no solo al aplicarlos sino también para producirlos. Así pues, se buscan microorganismos (que no sean descubiertos todavía) que puedan eliminar hongos sin ningún peligro. La universidad busca microorganismos en los restos de compost que originan los gusanos, algo que no se está buscando mucho actualmente, por lo que se puede decir que este proyecto de la universidad es original.
Este proyecto me gusta cada vez más conforme vamos avanzando, estamos descubriendo nuevas técnicas biotecnológicas (tanto las tradicionales como las nuevas) que nos permiten obtener muy buenos resultados. Nunca se sabe lo que haremos el día después XD.
Día 3, hoy nos hemos dedicado casi completamente a la extracción del DNA de los microorganismos. Para empezar teníamos que prepara lysozyma (que parece ser que se extrae de yema de huevo), pero por suerte ya estaba preparada, por lo que pudimos saltartnos este paso. Luego debíamos echar los microorganismos en un tubo pequeñito, y mezclarlo con el lysozyma, utilizando una máquina (el vórtice) en la cual simplemente había que colocar la punta del tubo sobre el vórtice y aguantar el tubo mientras la máquina vibraba y mezclaba los componentes. Seguidamente teníamos que colocar el tubo en otra máquina que permitiía mantenerlo a 37ºC, durante treinta minutos, tiempo que aprovechamos para almorzar.
Cuando ya estaban preparados los tubos, teníamos que echar a la mexcla unos 200 microlitros de proteína K, más otros 200 microlitros de «Lysis Solution», y mezclar, usando el vórtex, durante 15 segundos. Después teníamos que prepara unas columnas (que son unos tubos dentro de otros tubos) para echar la disolución resultante (tras haber debidamente lavado el tubo centrifugándolo). A esta disolución le echamos unos 200 microlitros de etanol y lo mezclamos con el vórtex durante unos diez segundos. Luego echamos el tubo en una centrifugadora, y cambiamos de recipiente para quedarnos con la columna y eliminar los restos originados. Esto lo volvimos a hacer dos veces, pero mezclando cada vez la disolución con una disolución de lavado. Por último, echamos 200 microlitros de «Elution Solution» en el centro de la columna y volvimos a centrifugar. Tras eliminar los restos, guardamos el DNA (que ya estaba bastante puro) en la «nevera».
 (pipeta)
(pipeta)  (procesos de la extracción)
(procesos de la extracción)
Este proceso nos ha llevado casi todo el tiempo del proyecto de hoy, y hasta hemos tenido que almorzar antes ya que todos los pasos deben estar perfectamente encadenados. Por suerte, Gonzalo nos había dado el día anterior unas hojas (en inglés) en las que ya ponía cómo iba a ser la extracción del DNA.
Al acabar esto, descansamos una media hora leyendo unos artículos (otra vez en inglés) sobre avances de biotecnología en el mundo actual. Pero rápidamente seguimos con nuestro proyecto: eb el poco tiempo que nos quedaba, teníamos que hacer un gel en el cual se pudiese incluír el ADN, para después examinarlo con luz ultravioleta. Pero hoy solo hicimos el gel, utilizando un líquido llamado TAE (que seguramente serán las iniciales de un nombre mucho más complicado), mezclándolo con una sustancia parecida al agar, y que curiosamente teníamos que apartarnos de la luz ya que la solución era sensible a ésta.
Pero hoy nos enfrentamos a un gran problema: no tenemos ni idea de cómo hacer la presentación de la semana que viene. Se nos había ocurrido hacer un par de vídeos en los que Borja y yo nos turnábamos para grabar los diferentes pasos del proyecto. Por desgracia, la cámara no era buena, y ninguno de nosotros teníamos ni idea de cómo manejar el vídeo en el ordenador, por lo que la duda quedaba aún en el aire.
Este es el penúltimo día del proyecto, lo cual es una pena porque me ha gustado mucho esta experiencia y he aprendido cosas muy interesantes, que me han ayudado a tener más claro mi futuro académico y laboral, a lo que me dedicaré y lo que estudiaré durante segundo de bachiller para acceder a la carrera que pienso hacer. Hoy no hemos avanzado mucho en el proyecto, hemos vuelto a hacer un gel (utilizando TAE, RED SAFE ( es un colorante) y agarosa (esta última hacía que el gel se volviese duro)), en el que hemos dejado unos huecos en los que hemos vertido el DNA que hemos obtenido junto a unos colorantes. El gel se metía en un líquido con sales, se le aplicaba una corriente eléctrica durante una media hora, y el DNA viajaba a través del gel desde la zona de carga negativa hacia la zona de carga positiva (recordar que el DNA es ácido desoxirribonucleico, y todos los ácidos tienen carga negativa, por lo que son atraídos por las cargas negativas).
 (electroforesis)
(electroforesis)
Después, colocamos el gel (con las marcas del viaje del DNA) en una cámara de luz ultravioleta, en la que se podía observar completamente el viaje del DNA y de los colorantes. No hemos podido hacer más.
Durante esa «media hora» que tardaba en hacerse la electroforesis (así se llama al procedimiento de aplicar una corriente eléctrica al gel), aprovechamos para hacer una vuelta por la politécnica y ver las demás instalaciones de biotecnología que había en ésta. Como estas instalaciones (denominadas CAMA, Centro Avanzado de Microbiología de Alimentos) estaba en la otra punta de la politécnica, tardabamos bastante más de una media hora, por eso no tuvimos tiempo de avanzar más en el proyecto.
ULTIMO DÍAAAA en la politécnica. Hoy realizamos la última parte del protecto: tras haber hecho Gonzalo una PCR (Reacción en Cadena de la Polimerasa, en inglés) de unos microorganismos y haberla mandado al centro de procesador, nos han enviado unas gráficas con los nucleótidos de dicho ADN. Solo tuvimos que, por ordenador, disponer la gráfica en distinto orden y pegar, ya que no es precisa en los extremos, por lo que la disponemos en dos sentidos para obtener una gráfica completa. Aquí confirmé algo que ya sabía, y es que soy malííííííísimo en informática, y en bioinformática también.
Nada más, nos dispunimos a hacer el vídeo para el polimedia y nos despedimos de los amigos y de Gonzalo (Gonzalo tambien es un amigo XD) lo que siempre da pena, sobre todo despues de haber tenido una experiencia tan agradable. Me ha encantado el Praktikum, tiene muchísmimas cosas positivas (y algunas negativas, como el polimedia) y me siento muy satisfecho de haber participado en esto.
Desde aquí un fuerte abrazo a mis colegas del Praktikum y todas las personas que nos han ayudado en este proyecto.